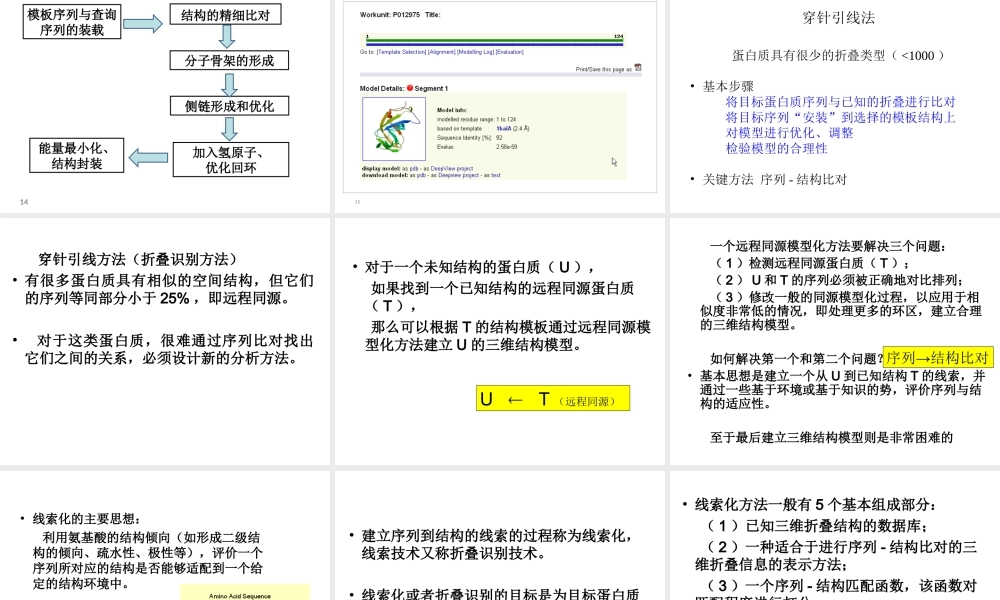
蛋白质结构预测肖飞蛋白质三级结构预测的方法123方法比较同源建模(比较建模)基础-相似的序列结构相近-PDB结构数据库的快速增长-结构基因组学的启动-发散进化特点-相对精确可靠•假设待预测三维结构的目标蛋白质为U(Unknown),利用同源模型化方法建立结构模型的过程包括下述6个步骤:(1)搜索结构模型的模板(T)(2)序列比对(3)建立骨架(4)构建目标蛋白质的侧链(5)构建目标蛋白质的环区(6)优化模型UT预测结果准确率:•对于具有60%等同的序列,用上述方法所建立的三维模型非常准确。若序列的等同部分超过60%,则预测结果将接近于实验得到的测试结果。•一般如果序列的等同部分大于30%,则可以期望得到比较好的预测结果。同源建模数据库搜索选择模板依据模板构建骨架模型环状、侧链的构建,优化结构合理性评估结构模型YESNO分子式:pGlu-His-Trp-Ser-Tyr-Gly-Leu-Arg-Pro-Gly-NH同源模建确定一对柔性分子相应功能团可能的空间取向模板加模板3.同源建模法的局限性传统的比较建模是通过PSI-BLAST找到已知结构的相关蛋白。最近如进行profile-profile比较和有效利用结构信息的更加复杂的方法已不仅显著增加了比对的质量而且远程同源(remotehomologue)检测的能力。因此,比较建模和折叠识别在基于模板的建模方法中的区别现已十分模糊。开发新的比较建模和折叠识别的算法导致网上各种预测方法的出现,这包括结构预测meta-服务器。蛋白质三维结构预测服务通过因特网对公众免费开放(同源建模):瑞士生物信息研究所SWISS-MODEL丹麦技术大学生物序列分析中心CPHmodels比利时拿摩大学ESyPred3D英国癌症研究中心3DJigsaw4.常用建模服务器和软件简介AccelrysDiscoveryStudio软件InsightIIFAMS同源蛋白质结构预测的方法1)片段组装法:SWISS-MODEL2)距离几何法:MODELLERSWISS-MODEL:http://swissmodel.expasy.org//SWISS-MODEL.htmlSWISS-MODEL:http://swissmodel.expasy.org//SWISS-MODEL.html提交查询序列14模板序列与查询序列的装载结构的精细比对分子骨架的形成侧链形成和优化加入氢原子、优化回环能量最小化、结构封装SWISS-MODEL的工作过程:15最后的预测结果蛋白质具有很少的折叠类型(<1000)•基本步骤将目标蛋白质序列与已知的折叠进行比对将目标序列“安装”到选择的模板结构上对模型进行优化、调整检验模型的合理性•关键方法序列-结构比对穿针引线法穿针引线方法(折叠识别方法)•有很多蛋白质具有相似的空间结构,但它们的序列等...